讲座|GEPIA数据库介绍
GEPIA(Gene Expression Profiling Interactive Analysis,基于基因表达水平值的交互式分析平台)是一个由北京大学张泽民教授团队开发的基因表达谱数据动态分析网页工具。该工具主要用于分析TCGA(The Cancer Genome Atlas)和GTEx(Genotype-Tissue Expression)项目中的RNA-seq表达数据。
是在线生信分析工具,无需数据下载,零代码操作。GEPIA中整理了每一个可检索的基因在不同肿瘤样本中的表达值,可以计算某个基因在某种肿瘤中的表达水平,还可以分析基因与肿瘤预后的关系、基因间的共表达等。
网址:http://gepia.cancer-pku.cn/ (GEPIA1.0版) 或 http://gepia2.cancer-pku.cn/ (GEPIA2.0版)。
GEPIA数据库功能概述
单基因分析(Single Gene Analysis):用户可以通过输入基因名称或Ensembl ID,获取该基因在不同肿瘤和正常样本中的表达情况。
癌症类型分析(Cancer Type Analysis):该功能包括差异基因分析(Differential Genes Analysis)和最显著生存基因(Most Significant Survival Genes)两部分。前者可以对选定癌种的差异基因进行分析,并在染色体上标示其位置;后者则汇总了与特定病种预后显著相关的基因。
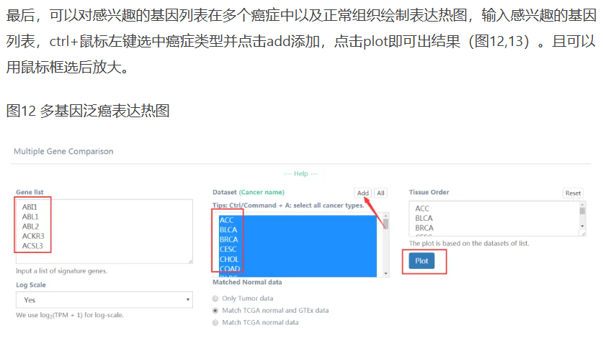
多基因分析(Multiple Gene Analysis):分为多基因比较(Multiple Gene Comparison)、相关性分析(Correlation Analysis)和降维分析(Dimensionality Reduction)三大功能。用户可以输入多个基因,选择肿瘤类型,进行表达分析、相关性分析和主成分分析(PCA),以便更好地理解基因间的关系和差异
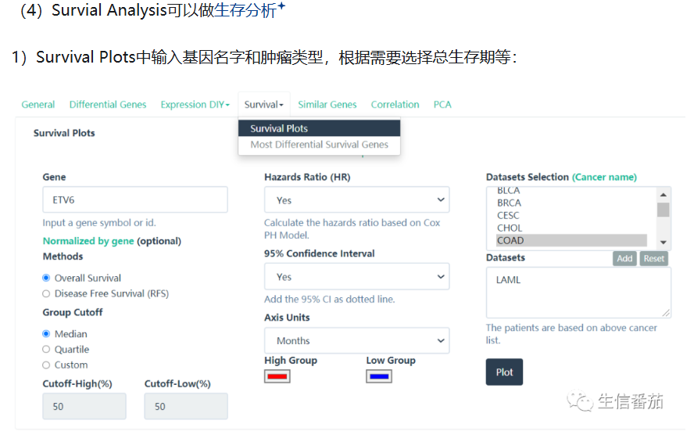
生存分析(Survival Analysis)GEPIA提供了高度自由的生存分析功能,用户可以输入基因名称,选择癌种,生成生存曲线图,并自动计算Logrank和HR值。该功能对于科研工作者和临床医生来说尤为重要
自定义数据分析(Custom Data Analysis)用户可以上传自己的肿瘤RNA-seq数据,与TCGA和GTEx数据进行比较,识别分子亚型、免疫亚型和泛癌亚型
详细使用教程参照:https://zhuanlan.zhihu.com/p/605320746,https://www.jianshu.com/p/fa850972f908
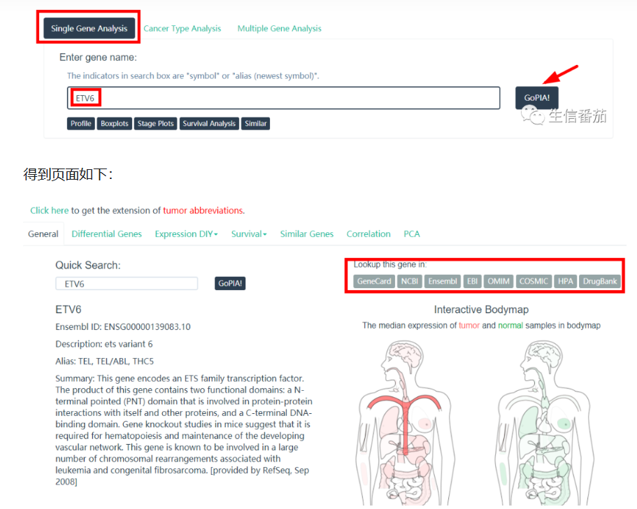
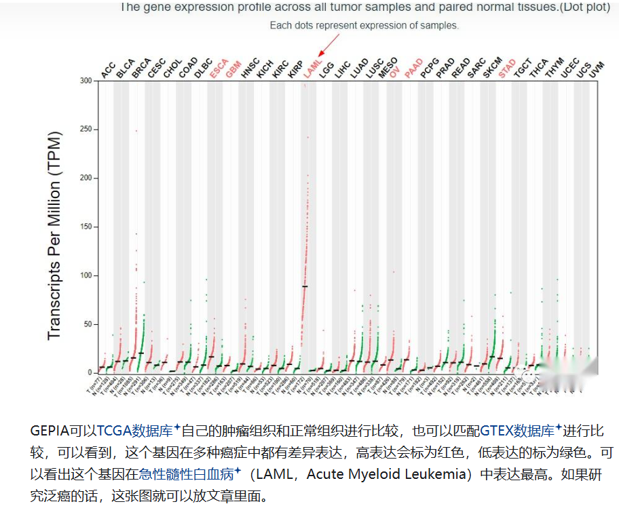
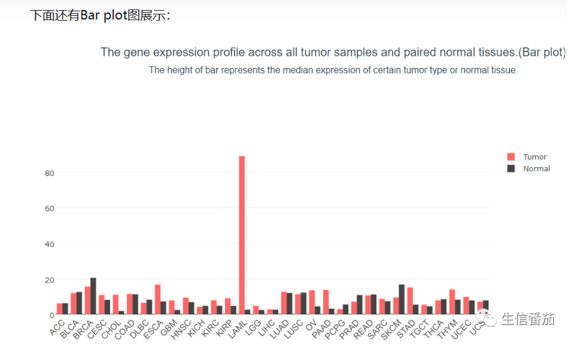
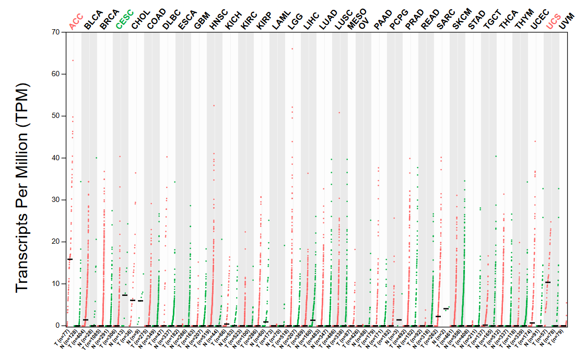
1)在搜索框中输入基因symbol或者基因名称,这里面输入了ETV6这个基因。两个示例人左边是tumor,右边是normal,颜色深浅分别代表高低表达,可以看到这个基因在血液肿瘤中的表达较其他组织深,而在其他组织中肉眼分不清楚到底是tumor中表达高还是在normal中表达高。另外这个基因也会与其他数据库关联,点击右上角可以跳转到其他数据库查看。
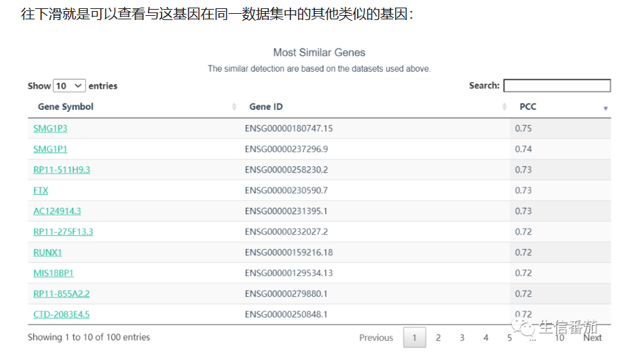
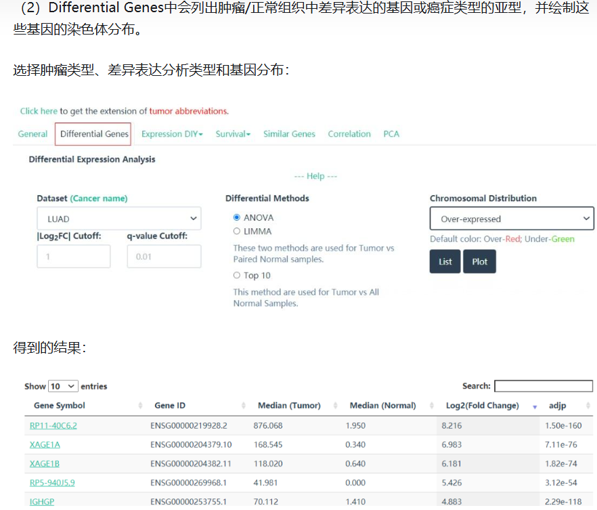
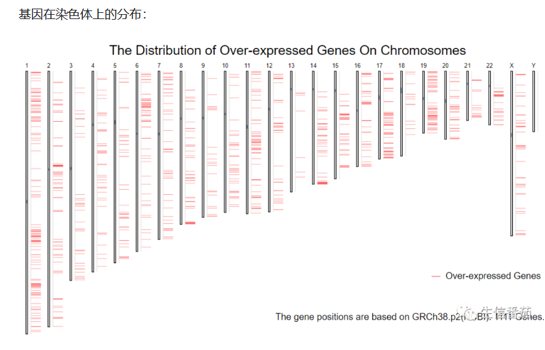
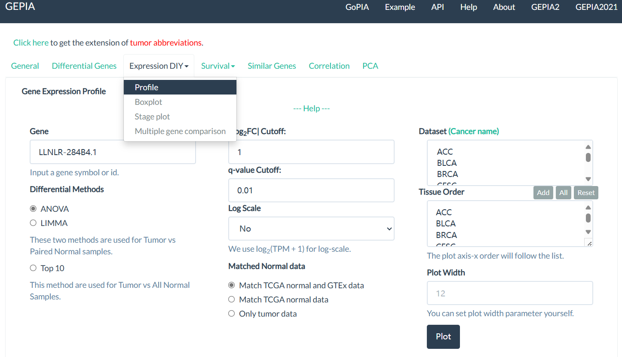
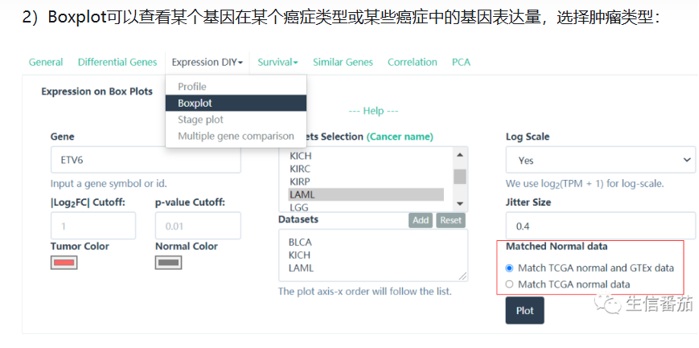
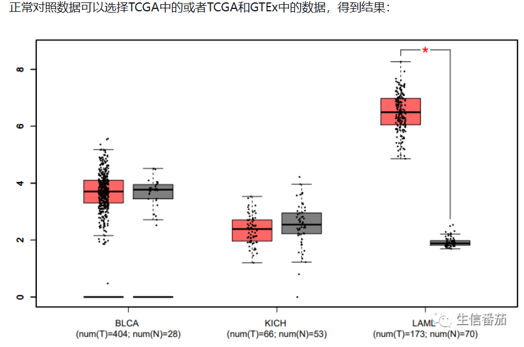
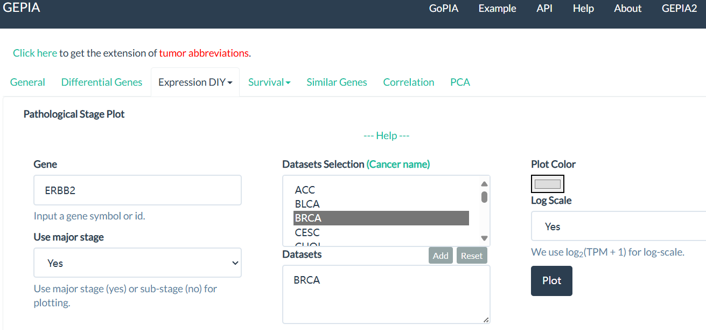
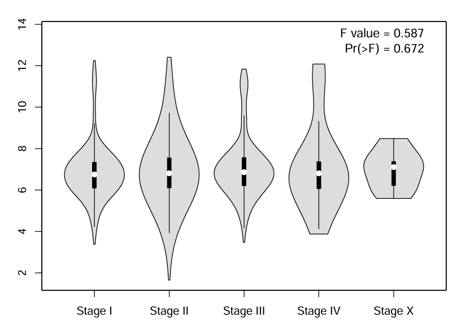
对于不同肿瘤分期,添加需要研究的肿瘤类型,同样可以对不同的分期中基因表达进行分析
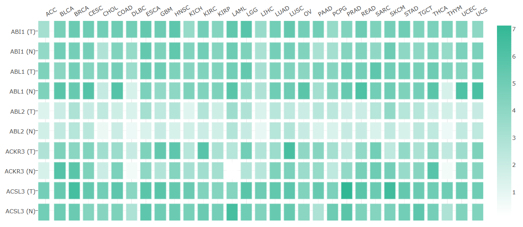
多基因泛癌表达分析
生存分析
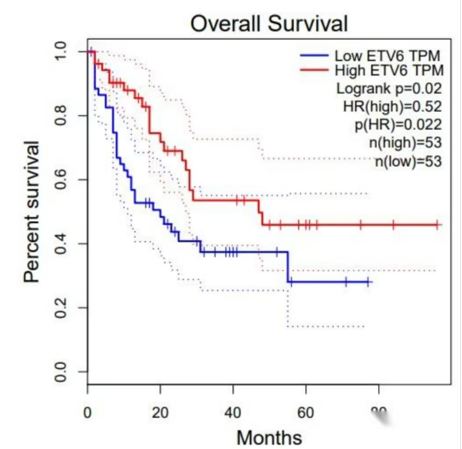
表达与预后有关的基因
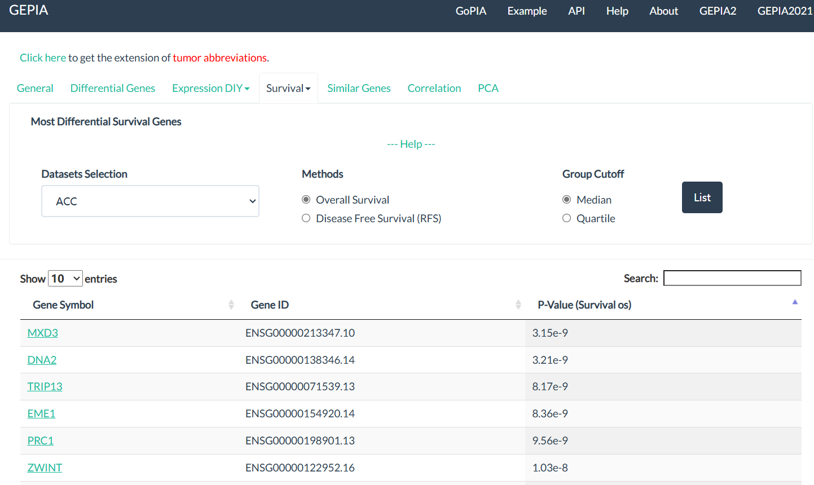
如果不知道癌症中哪些基因与生存有关,点击mostdifferential survival genes,得到与生存有关基因的列表,可以进行结果下载
基因之间的相关性分析
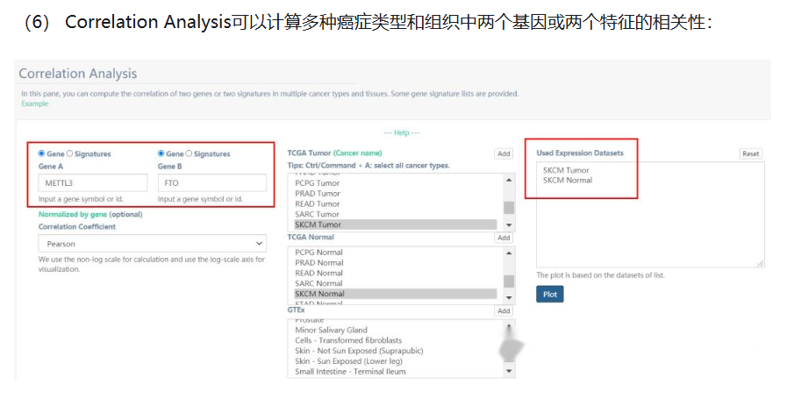
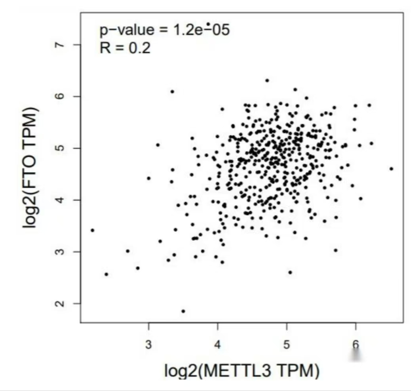
r为正值时,表示正相关。
r 为负值时,表示负相关。
r 越接近 1 或 -1,表示线性关系越强;接近 0 时,
表示没有明显的线性关系。
小结
TCGA数据库提供33种肿瘤的基因表达、基因突变、甲基化、临床信息等数据,数据下载后可通过工具进行分析,常用的分析工具如R语言(有很多不同功能的R语言包);
也有一些工具无需数据下载即可对TCGA的数据进行分析,如GEPIA,可在线对TCGA的数据进行分析(基因表达、生存分析等)。详细使用教程参照:https://zhuanlan.zhihu.com/p/605320746,https://www.jianshu.com/p/fa850972f908